北理工團隊利用3D重構(gòu)解析人類原腸運動早期發(fā)育過程
發(fā)布日期:2025-01-10 供稿:生命學院 攝影:生命學院
編輯:肖雯 審核:常非 閱讀次數(shù):
1月10日,北京理工大學肖振宇副教授、中國科學院動物研究所郭靖濤研究員、中國農(nóng)業(yè)大學魏育蕾教授、及中國科學院動物研究所于樂謙研究員合作在Nature Cell Biology上發(fā)表文章《Spatial Transcriptomic Characterization of a Carnegie Stage 7 Human Embryo》。這項研究將有助于進一步解讀人類早期原腸胚發(fā)育與細胞譜系特化的分子機制。北京理工大學副教授肖振宇為本文的共同通訊作者,碩士楊曉龍、博士謝鑫煒為共同第一作者。北京理工大學為共同通訊單位。
人的生命始于精子與卵子融合形成受精卵,進一步發(fā)育形成囊胚,之后經(jīng)過原腸運動起始早期器官形成的進程。人類原腸胚形成始于胚胎第14-15天(Carnegie stage,CS6-7),在約第21天(CS9)結(jié)束【1】,最終形成三胚層原腸胚。然而,早期原腸胚形成的研究仍較為有限。最近,同一研究團隊對人類CS8胚胎進行研究【2】,但許多早期原腸胚事件與細胞特化等已在更早階段發(fā)生。當前唯一可用的關于人類早期原腸胚的數(shù)據(jù)集是CS7階段的單細胞轉(zhuǎn)錄組數(shù)據(jù)【3】,但缺乏精確空間信息。人類早期原腸運動決定了細胞如何遷移、定位和分化,形成正常器官系統(tǒng),深入探討這些分子規(guī)律,為揭示發(fā)育異常和先天性畸形的根源提供理論基礎,提升對人類健康和疾病治療的理解。
為解決上述重大科學問題,該研究團隊基于CS7完整人類胚胎,利用連續(xù)橫斷面(transverse plane)高分辨空間轉(zhuǎn)錄組切片,結(jié)合深度學習算法進行三維對齊,重構(gòu)了首個帶有不同細胞類型與基因表達的數(shù)字3D人類原腸胚模型。
本工作結(jié)合空間位置與DEG對胚胎結(jié)構(gòu)細胞進一步分群,發(fā)現(xiàn)原腸運動原條細胞可細分出包括軸向中胚層、表達近軸中胚層、側(cè)板中胚層/胚外中胚層前體細胞等類群。此外,前內(nèi)臟內(nèi)胚層(Anterior Visceral Endoderm, AVE)的遷移行為對于確定胚胎的前后軸向至關重要,還具有限制原條在胚胎對側(cè)形成的能力【4】。本研究首次在人胚胎中證明了AVE的存在,并與鼠、猴等物種跨物種比較,發(fā)現(xiàn)其特征marker的保守性。
該研究還探討了人類原始生殖細胞(Primordial Germ Cells,PGCs)的空間分布與分子特性。通過與體內(nèi)PGCs、體外人類原始生殖細胞樣細胞(Human Primordial Germ Cell-like Cells,hPGCLCs)對比,驗證了CS7和CS8的PGCs在轉(zhuǎn)錄組特征上與hPGCLCs高度相似,進一步揭示了體內(nèi)和體外PGCs在發(fā)育過程中的分子變化。
鑒于3D模型難以論文圖片形式展示,為了便于更多讀者理解和使用研究結(jié)果,建立并發(fā)布了可互動共享網(wǎng)站:cs7.3dembryo.com. 這項研究推動了領域?qū)θ祟愒c胚發(fā)育的理解,并為未來早期人類發(fā)育研究提供了寶貴的數(shù)據(jù)集。
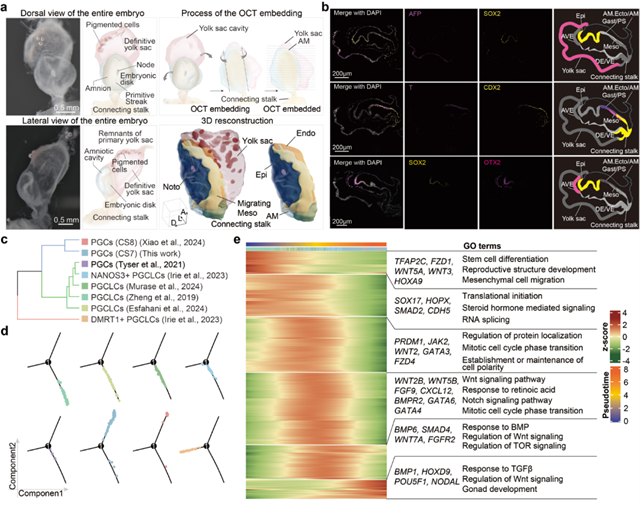
人類早期原腸運動胚胎結(jié)構(gòu)與譜系特化分析。
A. CS7胚胎空間轉(zhuǎn)錄組測序樣本形態(tài)與重構(gòu)模型。B. 免疫熒光染色驗證CS7胚胎胚層結(jié)構(gòu)。C. 結(jié)合體內(nèi)PGCs與hPGCLCs的Monocle2擬時序分析。D. C中不同細胞聚類關系的分支與相似程度。E. Monocle2進行PGCs模式基因富集的擬時序分析。
論文的第一單位為中科院動物所器官再生與智造重點實驗室,第三單位為北京理工大學生命學院。本研究得到國家重點研發(fā)計劃、國家自然科學基金委員會啟動資金等基金的資助。
原文鏈接:https://doi.org/10.1038/s41556-024-01597-3
1. Bergmann, S., Penfold, C. A., Slatery, E., Siriwardena, D., Drummer, C., Clark, S., Strawbridge, S. E., Kishimoto, K., Vickers, A., Tewary, M., Kohler, T. N., Hollfelder, F., Reik, W., Sasaki, E., Behr, R., & Boroviak, T. E. (2022). Spatial profiling of early primate gastrulation in utero. Nature , 609(7925), 136–143. https://doi.org/10.1038/s41586-022-04953-1.
2. Xiao, Z., Cui, L., Yuan, Y., He, N., Xie, X., Lin, S., Yang, X., Zhang, X., Shi, P., Wei, Z., Li, Y., Wang, H., Wang, X., Wei, Y., Guo, J., & Yu, L. (2024). 3D reconstruction of a gastrulating human embryo. Cell , 187(11), 2855–2874.e19. https://doi.org/10.1016/j.cell.2024.03.041.
3. Tyser, R. C. V., Mahammadov, E., Nakanoh, S., Vallier, L., Scialdone, A., & Srinivas, S. (2021). Single-cell transcriptomic characterization of a gastrulating human embryo. Nature , 600(7888), 285–289. https://doi.org/10.1038/s41586-021-04158-y.
4. Zhu Q, Ge J, Liu Y, Xu JW, Yan S, Zhou F. Decoding anterior-posterior axis emergence among mouse, monkey, and human embryos. Dev Cell . 2023 Jan 9;58(1):63-79.e4. doi: 10.1016/j.devcel.2022.12.004. PMID: 36626872.
分享到:
